건국대학교 KU융합과학기술원 김재범 교수(의생명공학과) 연구팀이 집단유전체 분석 프로그램인 ‘PAPipe’를 개발했다.
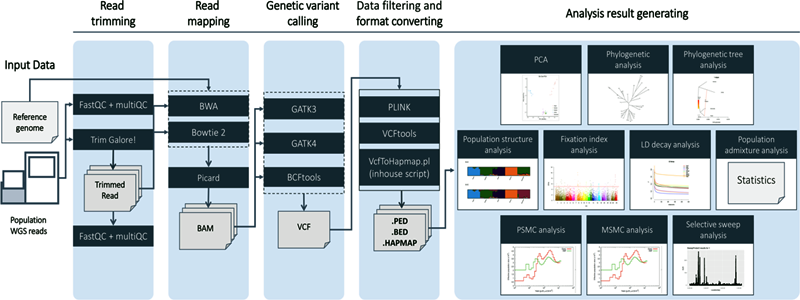
▲김재범 교수 연구팀이 개발한 집단유전체 분석 프로그램인 ‘PAPipe’를 활용하면 집단유전체 빅데이터 분석에 익숙하지 않은 많은 연구자들도 유전체 집단 분석을 쉽게 수행할 수 있다(PAPipe 데이터 분석 흐름도)
집단유전체 분석은 특정 집단을 구성하는 많은 개체의 방대한 양의 유전체를 상호 비교해 유전체 변이와 특이 형질 간의 연관성을 밝혀내는 작업이다. 그러나 분석해야 하는 유전체 데이터의 양 및 다양하고 많은 분석 과정과 그 복잡성 때문에 생물정보학에 익숙하지 않은 연구자들이 활용하기 어렵다.
이에 김재범 교수 연구팀이 개발한 ‘PAPipe’는 컴퓨터를 활용한 집단유전체 빅데이터 분석에 익숙하지 않은 많은 연구자들도 유전체 집단 분석을 쉽게 수행할 수 있도록 한다.
PAPipe를 활용하면 집단유전체 빅데이터에 대한 다양한 전처리 작업, 참조유전체에 대한 매핑 작업, 유전체 변이 발굴 작업, 그리고 이를 이용한 다양한 분석을 자동으로 수행할 수 있다. 이는 집단유전체 분석의 진입 장벽을 낮춰 많은 연구자들이 연구 대상 집단유전체의 다양한 특성을 탐색하고 이해하는데 크게 기여할 것으로 기대된다.
이번 연구 결과는 ‘Molecular Biology and Evolution(IF=10.7, 상위 5%)’에 게재됐으며, 생물학연구정보센터(BRIC)의 ‘한국을 빛내는 사람들’에도 소개됐다. 해당 논문의 제1저자는 건국대 의생명공학과 생물정보학연구실의 박나영 박사과정생이며, 이번 연구는 과학기술정보통신부의 지원으로 수행됐다.